SISTM
Les statistiques dans la biologie des systèmes et la médecine translationnelle


Objectifs
L’équipe se consacre au développement de méthodes statistiques pour l’analyse intégrative des données en médecine et en biologie. Grâce aux progrès technologiques, la recherche clinique et biologique génère des quantités massives de données. Les données » omiques » telles que la génomique (expression des gènes) et la protéomique sont importantes, mais aussi d’autres types de données, pour lesquelles les technologies modernes ont fortement augmenté la quantité d’informations (par exemple, l’imagerie médicale, les comptages cellulaires).
Cette équipe SISTM est labellisée à la fois par l’Inserm, l’université de Bordeaux et l’Inria.
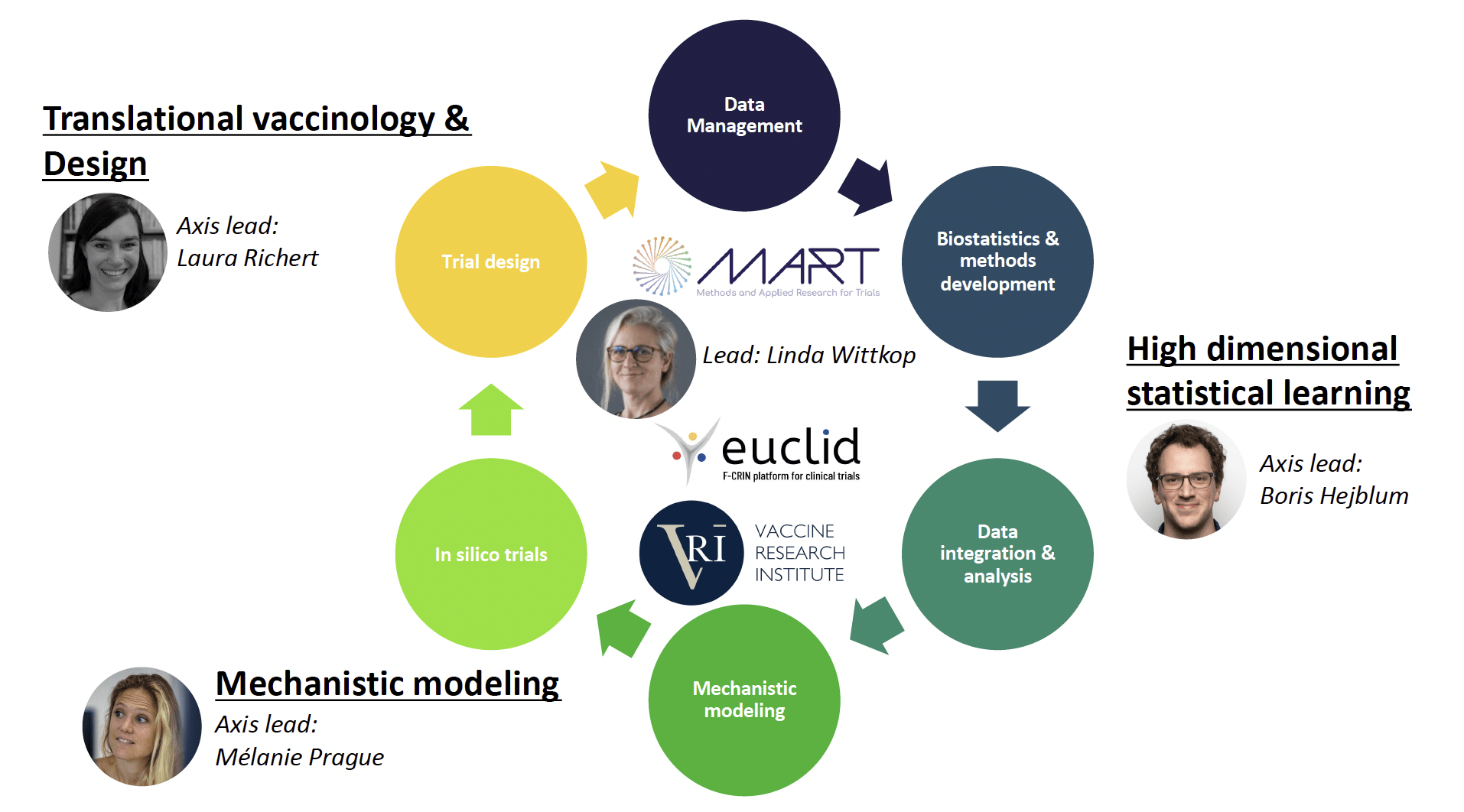
Les applications sont réalisées en collaboration avec l’Institut de recherche sur le vaccin (VRI), d’autres équipes du centre ainsi que l’Unité d’appui méthodologique à la recherche clinique et épidémiologique (USMR) du CHU de Bordeaux.
Les deux principaux objectifs de l’équipe SISTM sont :
-
- Accélérer le développement des vaccins en analysant toutes les informations disponibles lors des essais cliniques de phase précoce et en optimisant les nouveaux essais
- Développer de nouvelles méthodes statistiques pour analyser et modéliser les nombreuses données issues de mesures à haut débit.
Axes de recherche
En s’appuyant sur des connaissances biologiques extérieures, l’extraction d’informations pertinentes à partir de données omiques permet d’identifier un signal qui est ensuite intégré dans des modèles mécanistes afin d’en estimer les paramètres. Ces modèles peuvent ensuite orienter les stratégies vaccinales optimales à évaluer dans les prochains essais cliniques. De tels essais in silico, permettant alors de concevoir des essais cliniques optimisés avec des stratégies personnalisées, génèrent à leur tour de nouvelles données qui nécessitent et alimentent de nouveaux développements méthodologiques tels que ceux mentionnés précédemment.
Téléchargements
Pour l’estimation Lasso de la logistique conditionnelle.
Modèles de régression pour les études cas-témoins appariées et les études cas-croisés
Publications clés 2024
Alexandre M, Prague M, Lhomme E, Lelievre J-D, Wittkop L, Richert L, Levy Y, Thiebaut R. Definition of Virological Endpoints Improving the Design of HIV Cure Strategies Using Analytical Antiretroviral Treatment Interruption. Clin Infect Dis. 2024;79(6):1447-57. https://doi.org/10.1093/cid/ciae235
Barry H, Lhomme E, Surenaud M, Nouctara M, Robinson C, Bockstal V, Valea I, Somda S, Tinto H, Meda N, Greenwood B, Thiebaut R, Lacabaratz C. Helminth exposure and immune response to the two-dose heterologous Ad26.ZEBOV, MVA-BN-Filo Ebola vaccine regimen. PLoS Negl Trop Dis. 2024;18(4):e0011500. https://doi.org/10.1371/journal.pntd.0011500
Ganser I, Buckeridge DL, Heffernan J, Prague M, Thiebaut R. Estimating the population effectiveness of interventions against COVID-19 in France: A modelling study. Epidemics. 2024;46:100744. https://doi.org/10.1016/j.epidem.2024.100744
Hejblum BP, Ba K, Thiebaut R, Agniel D. Neglecting the impact of normalization in semi-synthetic RNA-seq data simulations generates artificial false positives. Genome Biol. 2024;25(1):281. https://doi.org/10.1186/s13059-024-03231-9
Hivert B, Agniel D, Thiébaut R, Hejblum BP. Post-clustering difference testing: Valid inference and practical considerations with applications to ecological and biological data. Comput Stat Data Anal. 2024;193:107916. https://doi.org/10.1016/j.csda.2023.107916
Levy Y, Moog C, Wiedemann A, Launay O, Candotti F, Hardel L, Durand M, […], Thiebaut R, Pantaleo G, Lelievre J-D, Richert L. Safety and immunogenicity of CD40.HIVRI.Env, a dendritic cell-based HIV vaccine, in healthy HIV-uninfected adults: a first-in-human randomized, placebo-controlled, dose-escalation study (ANRS VRI06). eClinicalMedicine. 2024;77:102845. https://doi.org/10.1016/j.eclinm.2024.102845
Loubet P, Lelievre J-D, Francois A, Botelho-Nevers E, Chidiac C, Chirio D, Dubee V, Dussol B, Galtier F, Hessamfar M, […], Launay O, Wittkop L. Humoral response after mRNA COVID-19 primary vaccination and single booster dose in people living with HIV compared to controls: A French nationwide multicenter cohort study-ANRS0001s COV-POPART. Int J Infect Dis. 2024;146:107110. https://doi.org/10.1016/j.ijid.2024.107110
Savel H, Meyer-Losic F, Proust-Lima C, Richert L. Statistical classification of treatment responses in mouse clinical trials for stratified medicine in oncology drug discovery. Sci Rep. 2024;14(1):934. https://doi.org/10.1038/s41598-023-51055-7
Segalas C, Helmer C, Genuer R, Proust-Lima C. Functional Principal Component Analysis as an Alternative to Mixed-Effect Models for Describing Sparse Repeated Measures in Presence of Missing Data. Stat Med. 2024;43(26):4899-912. https://doi.org/10.1002/sim.10214
Valayer S, Alexandre M, Prague M, Beavogui AH, Doumbia S, Kieh M, Greenwood B, Leigh B, Poupelin M, […], Thiebaut R, Levy Y, Yazdanpanah Y, Richert L, Lhomme E. Evaluation of waning of IgG antibody responses after rVSVDeltaG-ZEBOV-GP and Ad26.ZEBOV, MVA-BN-Filo Ebola virus disease vaccines: a modelling study from the PREVAC randomized trial. Emerg Microbes Infect. 2024;14(1):2432353. https://doi.org/10.1080/22221751.2024.2432353
Dernières actualités


Essai Clinique ANRS VRI06 de Phase I pour un vaccin préventif contre le VIH
presse
Un espoir dans la lutte contre le VIH : un candidat-vaccin préventif montre des résultats encourageants
presse
Organisation des journées de la statistique - Conférence annuelle de la Société Française de Statistique
Événements
Deux Chercheurs du BPH Obtiennent leur HDR : Boris Hejblum et Mélanie Prague
Awards
Lancement de CAP Santé numérique
dans le cadre de l’appel à manifestation d’intérêt
"Compétences et métiers d’avenir"
Événements

Covid-19 : Etude de l'efficacité des mesures instaurées contre le virus en France
presseMembres
Avalos FernandezMarta
Associate Professor in BiostatisticsBAKALIDOU
Etudiant en thèseBoespflugNicolas
Ingénieur d'étudesClaironQuentin
Chargé de Recherche INRIAColajanniAntonin
Etudiant en ThèseDarmignySandrine
Assistante administrativeEL HAJJZeinab
Ingénieure de rechercheEl KarchiJad
Ingénieur R&DFALLETSARA
Ingénieur d'étudesFerteThomas
Etudiant en thèseGABAUTAuriane
Étudiante en thèseGanserIris
Etudiante en thèseGounaDaniela
DoctoranteGUERRA ADAMESAriel
Doctoral StudentHejblumBoris
Chargé de Recherche INSERMHivertBenjamin
Post-doctorant / Post-doctoral fellowHuchonMélanie
Ingénieure d'études biostatisticienneHUGHESArthur
Etudiant en thèseLAVALQuentin
Ingénieur d'étudesLhommeEdouard
MD, PhD - MCU-PH Santé PubliqueLiZhe
DoctoranteNAFTI MAHERZIMyriam
Data ManagerOttaviAnton
Coordinateur de Projets InternationauxPalAnnesh
DoctorantPragueMélanie
Chargé de Recherche InriaREMIATJustine
Doctorante en Santé Publique - BiostatistiqueReynéBastien
PostdocRichertLaura
MD, PhD - PU-PH Santé PubliqueRuizAnne-Andrée
Ingénieure d'études BiostatisticienneSAVELHelene
Etudiante en thèseSegalasCorentin
Research Fellow in statisticsThiebautRodolphe
Directeur d'équipeWittkopLinda
MD PhD - PU-PH Santé Publique